Klassifikation der T-ALL
WGS und WTS können die genetische Diagnostik bereichern
Hintergund
Der genetische Hintergrund der T-ALL, die prognostische Bedeutung genetischer Veränderungen und deren potenzielle Auswirkung auf die Therapie wurden bereits in umfassenden Studien untersucht. Während bei der Aktualisierung der WHO-Klassifikation (5. Auflage, 2022) für die T-ALL jedoch noch keine genetisch definierten Gruppen eingeführt wurden, schlägt die „International Consensus Classification“ (ICC) acht Subgruppen als provisorische Entitäten vor. Für einen Schritt in Richtung personalisierter Medizin ist die spezifische Klassifizierung anhand leicht verfügbarer Biomarker unerlässlich. Eine Einteilung basierend auf genomischen Rearrangements lässt sich mit Standarddiagnostikverfahren vornehmen. Ob zudem Daten von Ganzgenom- und Ganztranskriptomsequenzierungen (WGS, WTS) zur Klassifizierung beitragen können, wurde im Rahmen eines Projektes evaluiert.
Ergebnisse
Mit Standarddiagnostikverfahren wurden 131 Patienten mit T-ALL untersucht. So konnten mittels Chromosomenbandenanalyse (CBA), FISH-Analysen für TRAD-, TRB-, TLX1-, TLX3-, NUP98- und HOXA9/10-Rearrangements sowie RT-PCRs zum Nachweis der Fusionen STIL::TAL1, SET::NUP214 und PICALM::MLLT10 60 der 131 T-ALL Patienten (46 %) neun genetischen Subgruppen zugeordnet werden: TLX1 (n = 22), TLX3 (n = 10), TAL1 (n = 4), HOXA9/10 (n = 4), SET::NUP214 (n = 7), MLLT10 (n = 4), NUP98 (n = 3), MYB (n = 2) und seltene Fusionen (LEF1-, LMO2- und NKX2-3-Rearrangements; n = 4) (Abb. 1). Zum Nachweis eines BCL11B::TLX3-Rearrangements sowie einer SET::NUP214- oder STIL::TAL1-Fusion sind FISH-Analysen bzw. molekulargenetische Analysen erforderlich, da es sich um zytogenetisch kryptische Veränderungen handelt, die bei der CBA nicht detektiert werden.
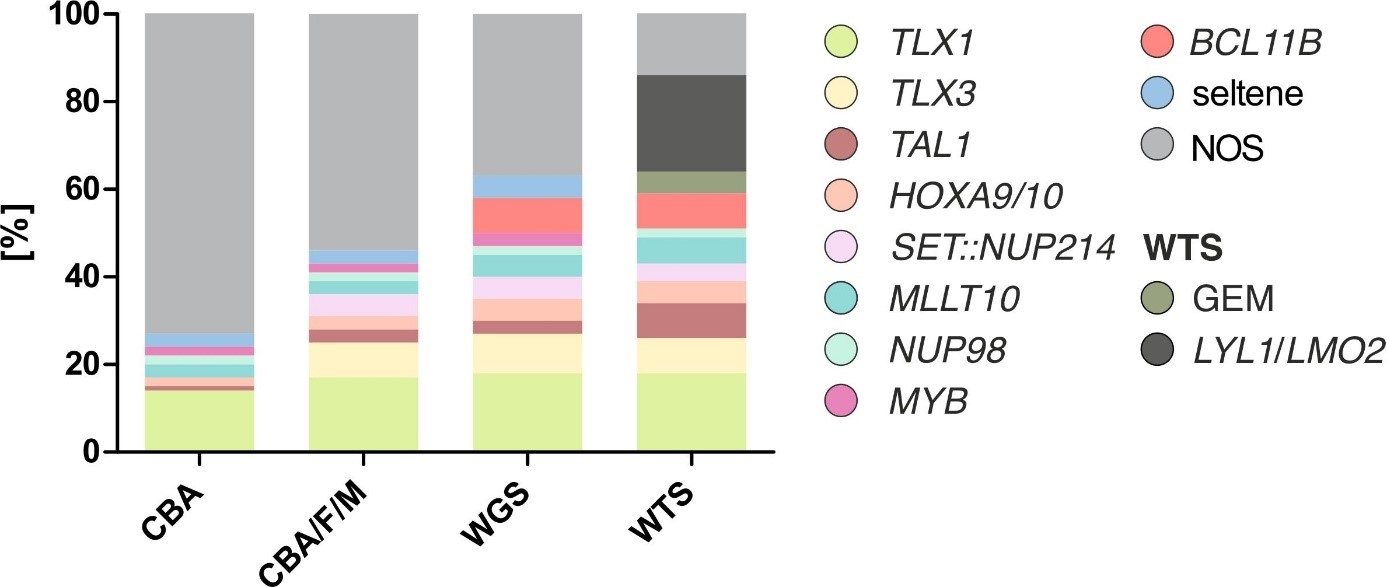
Zusätzlich konnten durch WGS-Analysen relevante Informationen für die Klassifizierung gewonnen werden. Bei 13 Patienten, bei denen entweder kein Material für FISH-Analysen (n = 4) oder keine FISH-Sonde bzw. RT-PCR für den Nachweis der jeweiligen Aberration (n = 9) verfügbar waren, konnten mittels WGS spezifische Rearrangements detektiert werden (CCDC26::TLX3, DDX3X::MLLT10, XPO1::MLLT10, TRB::MYB, TRB::NOTCH1; MYB-Enhancer-Mutation). Außerdem konnten zehn Fälle der kürzlich beschriebenen Subgruppe der BCL11B-Rearrangements zugeordnet werden (CCDC26::BCL11B (n = 2), ARID1B::BCL11B (n = 2), SATB1::BCL11B (n = 1) oder BCL11B-Enhancer-Amplifikation (n = 5)). BCL11B-Rearrangements wurden bei Patienten mit T-ALL, MPAL und undifferenzierter AML beschrieben. Da es sich hierbei ebenfalls um häufig zytogenetisch kryptische Veränderungen handelt, ist – solange WGS/WTS nicht Einzug in die Routinediagnostik erhalten – die Einführung einer BCL11B FISH-Sonde erforderlich, um T-ALLs der neuen WHO Entität „Acute leukaemia of ambiguous lineage with BCL11B rearrangement“ zuverlässig zu identifizieren.
Einschließlich der WGS-Daten konnten insgesamt 83 der 131 Patienten (63 %) einer spezifischen genetischen Subgruppe zugeordnet werden und nur 48 Fälle (37 %) wurden als "not otherwise specified (NOS)" bezeichnet. Dabei zeigten WTS-Daten bei der Mehrheit der NOS-Gruppe ein spezifisches Genexpressionsprofil mit Überexpression der Gene LYL1 und LMO2 (n = 27). Zudem wurde bei sieben Patienten ein ausgeprägtes Genexpressionsmuster (GEM) identifiziert, das durch eine hohe Expression von KCNG3, PTPRK und SCRN1 sowie eine geringe Expression von ERG, HOXA10, P2RY1, TTC28, ZBTB8A und ZNF618 gekennzeichnet war.
Somit konnte durch die Diagnostik mittels CBA, ergänzt durch ein FISH-Panel mit sechs Sonden und ein RT-PCR-Screening für drei Fusionen, eine Zuordnung von 46 % der T-ALL Patienten zu einer genetisch definierten Subgruppe erfolgen. Durch den Einschluss von WGS-Daten war es möglich insgesamt 63 % der Patienten basierend auf der Genetik zu klassifizieren und darüber hinaus konnten durch WTS-Analysen bei 34 Patienten zusätzliche Erkenntnisse gewonnen werden.